距离计算
聚类是“无监督学习”中研究最多,应用最广的一种。其特点是训练样本中的标记是未知的,通过无标记训练样本的学习来解释数据的内在性质和规律,将样本集划分为不相交的“簇”,并且簇内样本的相似性大于簇间样本的相似性。常见的聚类方式包括:原型(基于距离)聚类,密度聚类,和层次聚类。今天要实现的Kmeans算法就是一种典型的原型聚类算法。需要说明的是这几种聚类方式事实上都需要计算两个样本间的距离,只不过原型聚类直接以距离来表示两个样本的相似程度,所以如何表示样本间的距离是聚类里重很重要的一个问题,我先来总结一下常见的举例表示方式。
首先,规定函数\(dist(\cdot,\cdot)\),是一个“距离度量”,则它必须满足以下基本性质:
非负性:\(dist(x_i,x_j)\geq0\)
同一性:\(dist(x_i,x_j)=0\)当且仅当\(x_i=x_j\)
对称性:\(dist(x_i,x_j)=dist(x_j,x_i)\)
传递性:\(dist(x_i,x_j)\leq dist(x_i,x_k)+dist(x_k,x_j)\)
给定样本\(x_i=(x_{i1},x_{i2},…,x_{in})\)与\(x_j=(x_{j1},x_{j2},…,x_{jn})\),最常用的额是“闵可夫斯基”距离:
观察一下会发现这个距离其实就是在计算样本差的范数\(||x_i-x_j||_p\),我们在二维空间里常用的“欧式距离”就是当\(p=2\)时的“闵氏距离”:
当\(p=1\)时,也会得到一种比较著名的距离—曼哈顿距离(街区距离):
当然以上计算方式在连续属性里是比较常用的,而对于离散属性,如果这个离散属性是“有序”离散属性的话还是可以使用这种计算方式的,比如定义域为\({1,2,3}\)的某个属性。但是对于定义域为{北京,上海,广州}这样的定义域我们就不能直接使用以上距离方式了,因为在使用“闵可夫斯基距离”的时候没办法计算类似北京-上海这样的离散值距离。这里需要引入一种基于统计的距离计算方式—VDM法,它提供了一种用来计算某个离散属性上两种取值之间的差值,我们用\(m_{u,a}\)来表示属性u上取值为a的样本数,用\(m_{u,a,i}\)来表示第i个样本簇中在属性u上取值为a的样本数,k为样本簇的个数,则属性u上两个离散取值a,b之间的差值可以表示为:
值得注意的计算VDM距离需要知晓样本簇的划分情况,但是在聚类算法中,往往第一轮结束后才能得到一个簇的划分,算法刚开始的时候,样本簇完全是未知的,这个时候可以选择根据参数随机划分。现在假设样本的共有\(n\)个属性,其中前\(n_c\)个属性是连续的,后\(n-n_c\)个属性是离散且无序的,那么我们可以将\((1)\)式中的距离更新为如下形式:
当然,很多时候样本属性的权重是不同的,因此在计算样本距离的时候也要考虑到权重问题,并在计算距离的累加项中添加相应的权重因子\(w_i\)就行了,通常\(\sum_{i=1}^nw_i=1\)。
Kmeans聚类
需学习完距离表示之后,理解Kmeans就很简单了。给定训练样本\(D={x_1,x_2…x_m}\),算法的优化目标比较直接,就是对于所得的簇划分\(C={C_1,C_2…C_k}\),最小化其平方误差:
这里的\(\mu_i\)是第i个簇的均值向量,可以表示为:\(\mu_i=\frac{1}{|C_i|}\sum_{x\in C_i}x\),就是个简单的平均操作。
以上,我们可以将Kmeans的算法流程描述如下:
1.指定参数k,从样本集D中随机选出k个样本作为初始均值向量,也叫
核。2.迭代
计算各个样本和均值向量之间的距离,并将这个样本划分到与之距离最近的均值向量的簇中。
重新计算均值向量,如果均值向量有更新,则进行替换。
如果所有均值向量均为更新,则停止。
3.输出划分好的k个簇。
性能分析和参数选择
由于kmeans的迭代次数是不确定的,我们假设它为T,那么算法的时间复杂度实际上是:\(O(nkmT)\)其中:
T是迭代代价
k是遍历簇的代价
m是遍历簇中样本的代价
n是计算距离时遍历属性的代价
空间复杂度就比较简单了,只要能存下所有样本就行了,也就是一个m×n的矩阵:\(O(n*m)\)。
Kmeans算法常常需要解决的问题是:如何选取参数k以及如何选取初始均值向量。
关于k值的选择,参考书上提到一个规律:
给定一个合适的类簇指标,比如平均半径或直径,只要我们假设的类簇的数目等于或者高于真实的类簇的数目时,该指标上升会很缓慢,而一旦试图得到少于真实数目的类簇时,该指标会急剧上升。这里说的类簇的直径是指类簇内任意两点之间的最大距离,类簇的半径是指类簇内所有点到类簇中心距离的最大值。
也就是说,跟机器学习的很多算法一样,得调参来试。这个规律我暂时还没有测试验证,等以后熟练一下python的可视化工具后再开个博客总结一下吧。
关于初始均值向量的选择,参考书上给出了两个可行方案:
1.选择彼此距离尽可能远的K个点
比如首先随机选择一个点作为第一个初始类簇中心点,然后选择距离该点最远的那个点作为第二个初始类簇中心点,然后再选择距离前两个点的最近距离最大的点作为第三个初始类簇的中心点,以此类推,直至选出K个初始类簇中心点。
2.先对数据用层次聚类算法或者Canopy算法进行聚类,得到K个簇之后,从每个类簇中选择一个点,该点可以是该类簇的中心点,或者是距离类簇中心点最近的那个点。
Canopy算法简单描述:首先定义两个距离T1和T2,T1>T2,从初始的点的集合S中随机移除一个点P,然后对于还在S中的每个点I,计算该点I与点P的距离,如果距离小于T1,则将点I加入到点P所代表的Canopy中,如果距离小于T2,则将点I从集合S中移除,并将点I加入到点P所代表的Canopy中。迭代完一次之后,重新从集合S中随机选择一个点作为新的点P,然后重复执行以上步骤。
结合以上,还可以总结出Kmeans算法的缺点:
1.对于离群点和孤立点敏感;
2.k值选择;
3.初始聚类中心的选择;
4.只擅长发现球状簇。
python实现
这里我主要用到了python的矩阵工具包numpy和绘图工具包matplotlib,这两个东西直接用pip安装就行了,由于python是纯粹的解释性语言,所以目前我没见过python像java一样使用maven或者gradle这样的构建工具,不知道有没有。这两个包在做一些机器学习的算法时都挺常用的,尤其是numpy,里面有很多类似matlab里的矩阵和向量操作,比如计算范数一行就搞定了,很方便。但是这样毕竟是要牺牲速度的,所以可以选择先用numpy快速实现一个原型,然后再选择把算法过程化,或者用java和c++重构。
from numpy import *
import re
import matplotlib.pyplot as pl
# 计算欧式距离
def cacu_distance(sample1, sample2):
return sqrt(sum(power(sample2 - sample1, 2)))
# 随机初始化均值向量
def init_mu(data, k):
# 获取数据集的行列,即测例数目和特征维数
num, dim = data.shape
mu = zeros((k, dim))
cnt = 0;
choosed = []
while cnt < k:
choose = int(random.uniform(0, num))
if choose in choosed:
continue
else:
choosed.append(choose)
mu[cnt, :] = data[choose, :]
cnt += 1
return mu
# 迭代
def kmeans(data, k):
num = data.shape[0]
# zeros生成的是array,需要转换成matrix,注意这里的数组信息一定要用元组表示
cluster_info = mat(zeros((num, 2)))
mu_flag = True
mu = init_mu(data, k)
while mu_flag:
mu_flag = False
for i in range(num):
mindist = 100000.0
cluster = 0
for j in range(k):
dist = cacu_distance(mu[j, :],data[i, :])
if dist < mindist:
mindist = dist
cluster =j
if cluster_info[i, 0] != cluster:
mu_flag = True
cluster_info[i, :] = cluster,mindist**2
for i in range(k):
# 这句可能比较难理解,矩阵可以根据一个数组里的多个索引返回另一个矩阵。
points = data[nonzero(cluster_info[:, 0].A == i)[0]]
mu[i, :] = mean(points, axis=0)
return mu, cluster_info
# 可视化
def show_cluster(data, k, mu, cluster_info):
num, dim = data.shape
assert dim == 2
color_dict = {'0': 'r', '1': 'g', '2': 'k', '3': 'y', '4': 'b'}
assert k <= len(color_dict)
# mark data
for i in range(num):
cluster = int(cluster_info[i, 0])
if i == 0:
print(data[i, 0], data[i, 1])
pl.scatter(data[i, 0], data[i, 1], c=color_dict.get(str(cluster)), alpha=0.5)
# mark mu
label = ['a', 'b', 'c', 'd', 'e', 'f', 'g']
for i in range(k):
pl.scatter(mu[i, 0], mu[i, 1], c=color_dict.get(str(i)), s=20)
pl.show()
# 测试
if __name__ == '__main__':
data = []
print('loading data...')
data_file = open('E:/io/file.txt','r')
for line in data_file.readlines():
# 这里我很惊讶。python的string是不能用正则表达式split的
data_info = re.split('\s+', line.strip())
data.append([float(data_info[0]), float(data_info[1])])
print('loaded successfully...')
print('clustering...')
data = mat(data)
mu, cluster_info = kmeans(data,4)
print('finished...')
print(len(cluster_info))
show_cluster(data, 4, mu, cluster_info)
参数k=4的时候最终聚类结果图如下:
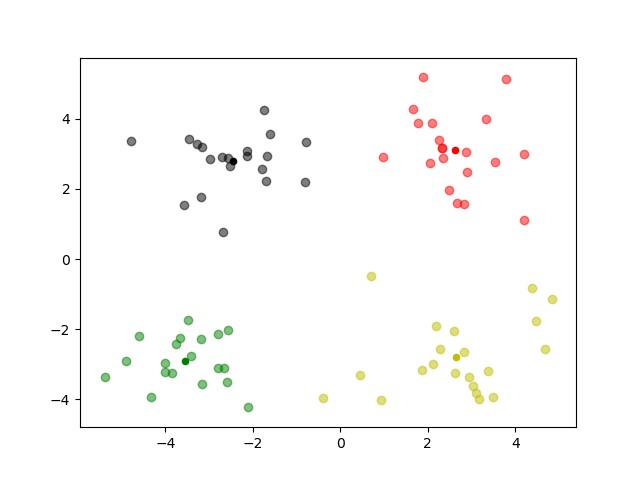
由于数据原因,这是个典型的球形聚类问题,所以不能反映kmeans的一些缺点,并且我没有对参数选择的性能进行详细测试,以后有时间再弄吧。
java实现
以前上数据挖掘的时候用java实现的一个代码,比较low,但是还是放上来吧,这段代码绝对是要比python实现速度快很多的:
import java.io.BufferedWriter;
import java.io.FileWriter;
import java.util.Random;
import java.util.Scanner;
class point {
public float x = 0;
public float y = 0;
public int flage = -1;
public float getX() {
return x;
}
public void setX(float x) {
this.x = x;
}
public float getY() {
return y;
}
public void setY(float y) {
this.y = y;
}
}
public class KMeans {
int time = 1;
point[] ypo;
point[] pacore = null;
point[] pacoren = null;
String info;
static BufferedWriter output = null;
static String path = "F://File//result.txt";
public void productpoint() throws Exception{
Scanner cina = new Scanner(System.in);
System.out.print("请输入聚类中点的个数(随机产生):");
int num = cina.nextInt();
ypo = new point[num];
for (int i = 0; i < num; i++) {
float x = (int) (new Random().nextInt(10));
float y = (int) (new Random().nextInt(10));
ypo[i] = new point();
ypo[i].setX(x);
ypo[i].setY(y);
}
output.write("共产生了"+num+"个数据,以下是详细信息:\n");
for(int i=0; i<num; i++){
output.write(ypo[i].x+","+ypo[i].y+"\n");
}
output.write("\n");
System.out.print("请输入初始化聚类中心个数(随机产生):");
int core = cina.nextInt();
this.pacore = new point[core];
this.pacoren = new point[core];
Random rand = new Random();
int temp[] = new int[core];
temp[0] = rand.nextInt(num);
pacore[0] = new point();
pacore[0].x = ypo[temp[0]].x;
pacore[0].y = ypo[temp[0]].y;
pacore[0].flage=0 ;
for (int i = 1; i < core; i++) {
int flage = 0;
int thistemp = rand.nextInt(num);
for (int j = 0; j < i; j++) {
if (temp[j] == thistemp) {
flage = 1;
break;
}
}
if (flage == 1) {
i--;
} else {
pacore[i] = new point();
pacore[i].x= ypo[thistemp].x;
pacore[i].y = ypo[thistemp].y;
pacore[i].flage = 0;
}
}
output.write("共产生了"+core+"个中心,以下是详细信息:\n");
for(int i=0; i<core; i++){
output.write(pacore[i].x+","+pacore[i].y+"\n");
}
output.write("\n");
}
public void searchbelong()
{
for (int i = 0; i < ypo.length; i++) {
double dist = 999;
int lable = -1;
for (int j = 0; j < pacore.length; j++) {
double distance = distpoint(ypo[i], pacore[j]);
if (distance < dist) {
dist = distance;
lable = j;
}
}
ypo[i].flage = lable + 1;
}
}
public void calaverage() throws Exception{
output.write("以下是第"+time+"次迭代结果:\n");
output.write("\n");
for (int i = 0; i < pacore.length; i++) {
output.write("以<" + pacore[i].x + "," + pacore[i].y + ">为质心的点有:\n");
int numc = 0;
point newcore = new point();
for (int j = 0; j < ypo.length; j++) {
if (ypo[j].flage == (i + 1)) {
output.write(ypo[j].x + "," + ypo[j].y+"\n");
numc += 1;
newcore.x += ypo[j].x;
newcore.y += ypo[j].y;
}
}
pacoren[i] = new point();
pacoren[i].x = newcore.x / numc;
pacoren[i].y = newcore.y / numc;
pacoren[i].flage = 0;
}
}
public double distpoint(point px, point py) {
return Math.sqrt(Math.pow((px.x - py.x), 2) + Math.pow((px.y - py.y), 2));
}
public void change_oldtonew(point[] old, point[] news) throws Exception{
for (int i = 0; i < old.length; i++) {
old[i].x = news[i].x;
old[i].y = news[i].y;
old[i].flage = 0;
}
output.write("不满足要求,对聚类中心进行调整后的质心为:\n");
for (int i = 0; i < old.length; i++) {
output.write(old[i].x+","+old[i].y+"\n");
}
output.write("\n");
}
public void movecore() throws Exception{
this.searchbelong();
this.calaverage();
double movedistance = 0;
int biao = -1;
for (int i = 0; i < pacore.length; i++) {
movedistance = distpoint(pacore[i], pacoren[i]);
if (movedistance < 0.001) {
biao = 0;
} else {
biao=1;
break;
}
}
if (biao == 0) {
System.out.println("迭代完成,共进行了"+time+"次迭代!\n");
output.write("迭代完毕!,共进行了"+time+"次迭代。\n");
} else {
time++;
change_oldtonew(pacore, pacoren);
movecore();
}
}
public static void main(String[] args) throws Exception{
KMeans kmean = new KMeans();
output = new BufferedWriter(new FileWriter(path,false));
kmean.productpoint();
kmean.movecore();
output.flush();
output.close();
}
}